椎间盘退行性变(IDD)是指在多种病因作用下导致椎间盘生物力学和组织结构改变、髓核水分减少、纤维环破裂、压迫脊髓和神经根进而引起腰腿痛的疾病,近年来逐渐趋于年轻化[1-2]。IDD是脊柱外科的研究重点之一,但是其病因和发生机制尚不明确。有研究[3-7]报道,IDD主要与炎性反应、细胞衰老和细胞外基质成分改变等有关。因此,深入探索IDD的发生机制、寻找其早期诊断标志物和治疗靶点具有重要意义。近年来,随着生物信息学技术的发展和普及,许多疾病相关的基因组测序成为研究热点。本研究通过基因表达汇编(GEO)数据库中IDD相关基因芯片数据,分析筛选获取差异表达基因(DEG),并对DEG进行功能富集和蛋白质-蛋白质相互作用(PPI)网络分析,以挖掘IDD疾病的新型标志物,为IDD的早期诊治提供新的思路。
1 材料与方法 1.1 数据收集从GEO中搜索脊柱IDD相关的芯片数据。下载GSE124272[8]和GSE150408[9]数据集中的芯片数据,平台文件均为GPL21185(Agilent-072363 SurePrint G3 Human GE v3 8x60K Microarray 039494)。GSE124272中包含健康志愿者和IDD患者外周全血样本各8例,GSE150408中包含健康志愿者和IDD患者外周全血样本各17例。下载GSE23130[10]数据集中的芯片数据作为验证集,平台文件为GPL1352[(U133_X3P)Affymetrix Human X3P Array],依据Thompson退行性变等级[11]分类将23例总样本分为正常纤维环样本15例和IDD纤维环样本8例。
1.2 数据校正将GSE124272和GSE150408芯片数据整合,并通过平台文件中的基因名称对芯片数据进行注释。使用R 4.0软件sva数据分析包对2个芯片数据进行校正,去除批次效应,达到联合分析的目的。
1.3 筛选DEG通过R 4.0软件limma数据分析包比较正常人和IDD患者外周血中的基因表达改变。DEG的筛选标准均设定为校正后P<0.05和| log2差异倍数|≥0.585,绘制火山图。然后依据DEG在不同样本中的表达情况进行聚类分析,观察基因间和样本间的分布关系。
1.4 基因功能和通路富集分析基因本体(GO)是用于基因注释和分析基因生物学过程的主要生物信息学工具,京都基因与基因组百科全书(KEGG)是用于了解高级功能和生物系统的大规模分子数据库。使用在线数据库DAVID(https://david.ncifcrf.gov/)对DEG进行GO和KEGG通路分析,设定P<0.05为有统计学意义。
1.5 PPI网络分析将DEG导入STRING(http://string-db.org/)在线分析网站,按照组合得分>0.9的标准并隐藏未参与构建PPI网络的蛋白,将输出结果导入Cytoscape 3.7.1软件进行可视化。利用Cytoscape 3.7.1软件的MCODE插件筛选PPI网络中最为显著的蛋白模块获取关键基因,并观察其在验证数据集中的表达情况。
1.6 关键基因对IDD的诊断价值分析利用GSE124272、GSE150408数据集中的数据,通过计算受试者工作特征(ROC)曲线评估关键基因在IDD中表达的诊断价值,曲线下面积即代表基因的诊断效能。
2 结果 2.1 DEG筛选结果通过将GSE124272和GSE150408芯片数据联合分析后,共筛选出DEG 597个,包含上调基因363个和下调基因234个(图 1)。聚类分析将表达量相近的基因聚集,结果显示4组样本质量合格,并且无批次效应的影响。GSE23130数据集经分析后,共筛选出DEG 1 017个。

|
图 1 DEG筛选 Fig. 1 Screening of DEG 注:红点表示上调基因,绿点表示下调基因。筛选标准为基因表达改变量大于或等于1.5倍(| log2差异倍数|≥0.585),且P<0.05。 Note: Red dots indicate up-regulated genes, and green dots indicate down-regulated genes. Screening criteria is that amounts of gene expression change are greater than or equal to 1.5 times(| log2 fold change |≥0.585), and P < 0.05. |
GO功能分析分为生物过程、细胞组分和分子功能3个部分,分析结果显示,DEG参与的生物过程主要为细胞黏附、生物黏附和细胞间黏附等,细胞组分主要为褶皱膜、细胞间连接和核质等,而分子功能则以多聚RNA结合、蛋白酶体结合和蛋白复合物结合等为主(图 2a),提示DEG主要参与细胞黏附、细胞凋亡、趋化作用和细胞迁移等功能。KEGG信号通路分析结果显示,DEG主要参与细胞外基质受体相互作用和癌症中的信号通路(图 2b)。

|
图 2 DEG的GO和KEGG分析 Fig. 2 GO and KEGG analysis of DEG a:GO分析 b:KEGG分析 a: GO analysis b: KEGG analysis |
由图 3可见,PPI网络共有171个节点(蛋白)和313条边(蛋白之间的相互联系)。利用MCODE插件进行蛋白模块分析,获得最为显著的2个模块和17个关键基因,模块1由9个节点(LSM2、POLR2F、RBMX、POLR2C、HNRNPD、SUGP1、SRRT、SRSF7和PPIL4)和36条边组成(图 4a),模块2由8个节点(RPS3A、RPL15、SSR1、RPL23A、RPS29、EEF1A1、SMG1和RPL22)和25条边构成(图 4b)。经过GSE23130数据集验证发现,RBMX、EEF1A1、SSR1和POLR2C在外周血和椎间盘组织样本中均为DEG(图 4c)。
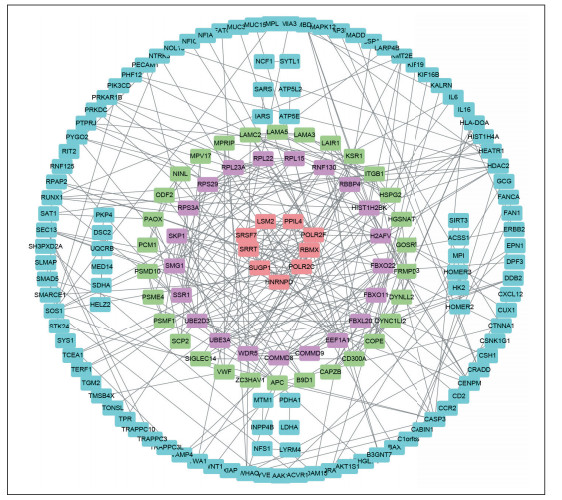
|
图 3 PPI网络分析 Fig. 3 PPI network analysis |
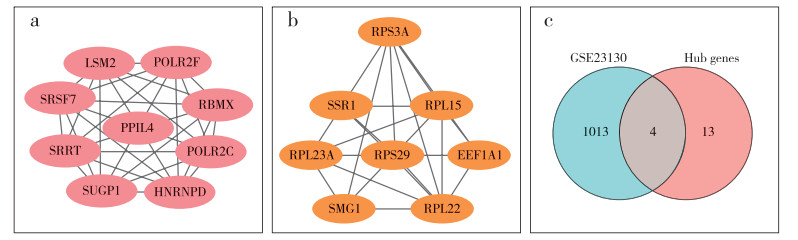
|
图 4 模块1、2的PPI网络分析及关键基因验证 Fig. 4 PPI network analysis and hub gene verification of modules 1, 2 a:模块1,借助MCODE插件分析获得PPI网络中最显著的模块,包含9个关键基因 b:模块2,借助MCODE插件分析获得PPI网络中第二显著模块,包含8个关键基因 c:通过GSE23130数据集验证发现2个模块共有4个交集基因 a: Module 1, most significant module in PPI network is obtained through MCODE analysis, which contains 9 hub genes b: Module 2, second significant module in PPI network is obtained through MCODE analysis, which contains 8 hub genes c: There are 4 overlapping genes between 2 modules through verification of GSE23130 dataset |
ROC曲线分析结果显示,外周血中RBMX、EEF1A1、SSR1和POLR2C对IDD诊断均具有一定价值,曲线下面积分别为0.763、0.741、0.710、0.702(图 5a~d),4个基因构建的联合诊断模型诊断价值进一步提高(曲线下面积为0.795,图 5e),提示这4个基因可作为IDD诊断的血液学标志物。
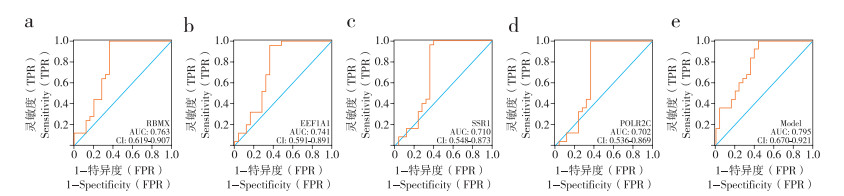
|
图 5 关键基因的诊断能力 Fig. 5 Diagnostic efficacy of hub genes a:RBMX的诊断效能 b:EEF1A1的诊断效能 c:SSR1的诊断效能 d:POLR2C的诊断效能 e:4个基因联合诊断效能 a: Diagnostic efficacy of RBMX b: Diagnostic efficacy of EEF1A1 c: Diagnostic efficacy of SSR1 d: Diagnostic efficacy of POLR2C e: Combined diagnostic efficacy of 4 genes |
IDD是临床常见病和多发病,严重影响患者生活质量。因此,通过生物信息学技术探索IDD的发生机制,挖掘疾病相关的生物标志物,对IDD的早期诊治具有重要意义。本研究结果表明,DEG主要参与细胞黏附、细胞凋亡、趋化作用和细胞迁移等功能。有研究[12]表明,在细胞和动物实验中通过抑制凋亡表型,可防止椎间盘过早发生退行性变,使其继续维持正常的生理功能,这或将成为IDD的治疗方向之一。本研究的KEGG信号通路富集分析结果显示,DEG主要富集于细胞外基质受体相互作用和癌症中的信号通路。并在分析过程中引入包含椎间盘组织样本的GSE23130数据集来进一步佐证结果的科学性和准确性,筛选出RBMX、EEF1A1、SSR1和POLR2C 4个关键基因,通过PPI网络分析和ROC曲线分析进一步明确了这4个关键基因可作为IDD的诊断标志物。
RBMX基因是着丝粒非编码RNP复合体的一个组成部分,其表达与Caspase3相关,并参与损伤后神经节细胞的凋亡过程[13]。EEF1A1负责将氨基酰tRNA酶解到核糖体,有研究[14-15]发现,EEF1A1可有效保护帕金森疾病引起的脑神经退行性变和死亡,与EEF1A2亚型共同参与神经退行性变性的进展。SSR1是一种糖基化内质网膜受体,与通过内质网膜的蛋白易位有关,可作为多种癌症的预后指标[16]。POLR2C编码RNA聚合酶Ⅱ的第三大亚基;Zhu等[17]的研究发现,POLR2C为腰椎IDD的关键基因,可作为IDD治疗的重要靶点。上述研究结果进一步支持了本研究结果的可信度。
全血组织样本获取途径极为便捷,筛选血液学诊断标志物可为疾病的早期诊治提供方向。Kyritsis等[18]分析脊髓损伤患者急性期外周血整体基因表达,鉴别急性脊髓损伤后全血细胞中自然杀伤细胞和巨噬细胞等的变化,后构建损伤程度预测模型,依据预测效力筛选出对脊髓损伤严重程度具有诊断和预后价值的生物标志物。Grad等[19]首次发现血浆中CCL5和CXCL6的升高与IDD程度密切相关,并推测这些趋化因子可能是诊断和监测IDD的血液学生物标志物。Qi等[20]证实,血清CTX-Ⅱ和COMP是诊断IDD的可靠指标,其浓度与IDD的发生过程呈正相关。本研究通过生物信息学分析方法筛选出IDD患者外周血诊断标志物RBMX、EEF1A1、SSR1和POLR2C,四者联合应用可提高对IDD的诊断效力。通过生物信息学方法筛选诊断标志物对IDD的早期诊治具有重大临床意义,也为后期实验提供了新的研究策略。
综上所述,通过生物信息学技术联合分析基因表达汇编数据芯片,探索IDD的诊断标志物,为进一步阐明IDD的发生机制及早期临床诊治提供理论参考,并为IDD靶向治疗药物的研发提供方向。
| [1] |
Sinkemani A, Wang F, Xie Z, et al. Nucleus pulposus cell conditioned medium promotes mesenchymal stem cell differentiation into nucleus pulposus-like cells under hypoxic conditions[J]. Stem Cells Int, 2020, 2020: 8882549. |
| [2] |
Liu L, He J, Liu C, et al. Cartilage intermediate layer protein affects the progression of intervertebral disc degeneration by regulating the extracellular microenvironment(review)[J]. Int J Mol Med, 2021, 47(2): 475-484. |
| [3] |
Liu ZM, Lu CC, Shen PC, et al. Suramin attenuates intervertebral disc degeneration by inhibiting NF-κB signalling pathway[J]. Bone Joint Res, 2021, 10(8): 498-513. DOI:10.1302/2046-3758.108.BJR-2020-0041.R3 |
| [4] |
Liao Z, Li S, Liu R, et al. Autophagic degradation of gasdermin d protects against nucleus pulposus cell pyroptosis and retards intervertebral disc degeneration in vivo[J]. Oxid Med Cell Longev, 2021, 2021: 5584447. |
| [5] |
Yan Z, Pan Y, Wang S, et al. Static compression induces ecm remodeling and integrin α2β1 expression and signaling in a rat tail caudal intervertebral disc degeneration model[J]. Spine(Phila Pa 1976), 2017, 42(8): E448-E458. DOI:10.1097/BRS.0000000000001856 |
| [6] |
袁宇飞, 彭宝淦. 腰椎椎间盘退变过程中炎症介质作用的研究进展[J]. 脊柱外科杂志, 2012, 10(5): 310-314. DOI:10.3969/j.issn.1672-2957.2012.05.015 |
| [7] |
张威林, 王海强, 陈宇飞, 等. 椎间盘退变的分子机制研究现状[J]. 脊柱外科杂志, 2012, 10(4): 253-256. DOI:10.3969/j.issn.1672-2957.2012.04.016 |
| [8] |
Wang Y, Dai G, Li L, et al. Transcriptome signatures reveal candidate key genes in the whole blood of patients with lumbar disc prolapse[J]. Exp Ther Med, 2019, 18(6): 4591-4602. |
| [9] |
Wang Y, Dai G, Jiang L, et al. Microarray analysis reveals an inflammatory transcriptomic signature in peripheral blood for sciatica[J]. BMC Neurol, 2021, 21(1): 50. DOI:10.1186/s12883-021-02078-y |
| [10] |
Gruber HE, Hoelscher GL, Ingram JA, et al. Genome-wide analysis of pain-, nerve- and neurotrophin-related gene expression in the degenerating human annulus[J]. Mol Pain, 2012, 8: 63. |
| [11] |
Thompson JP, Pearce RH, Schechter MT, et al. Preliminary evaluation of a scheme for grading the gross morphology of the human intervertebral disc[J]. Spine(Phila Pa 1976), 1990, 15(5): 411-415. DOI:10.1097/00007632-199005000-00012 |
| [12] |
Yokozeki Y, Kawakubo A, Miyagi M, et al. Reduced TGF-βexpression and CD206-positive resident macrophages in the intervertebral discs of aged mice[J]. Biomed Res Int, 2021, 2021: 7988320. |
| [13] |
Dai M, Liu Y, Nie X, et al. Expression of RBMX in the light-induced damage of rat retina in vivo[J]. Cell Mol Neurobiol, 2015, 35(4): 463-471. DOI:10.1007/s10571-014-0140-z |
| [14] |
Chalorak P, Dharmasaroja P, Meemon K. Downregulation of eEF1A/EFT3-4 enhances dopaminergic neurodegeneration after 6-OHDA exposure in C. elegans model[J]. Front Neurosci, 2020, 14: 303. DOI:10.3389/fnins.2020.00303 |
| [15] |
Abbott CM, Newbery HJ, Squires CE, et al. eEF1A2 and neuronal degeneration[J]. Biochem Soc Trans, 2009, 37(Pt 6): 1293-1297. |
| [16] |
Yan J, Wang ZH, Yan Y, et al. RP11-156L14.1 regulates SSR1 expression by competitively binding to miR-548ao-3p in hypopharyngeal squamous cell carcinoma[J]. Oncol Rep, 2020, 44(5): 2080-2092. |
| [17] |
Zhu J, Zhang X, Gao W, et al. lncRNA/circRNA-miRNA-mRNA ceRNA network in lumbar intervertebral disc degeneration[J]. Mol Med Rep, 2019, 20(4): 3160-3174. |
| [18] |
Kyritsis N, Torres-Espin A, Schupp PG, et al. Diagnostic blood RNA profiles for human acute spinal cord injury[J]. J Exp Med, 2021, 218(3): e20201795. DOI:10.1084/jem.20201795 |
| [19] |
Grad S, Bow C, Karppinen J, et al. Systemic blood plasma CCL5 and CXCL6:potential biomarkers for human lumbar disc degeneration[J]. Eur Cell Mater, 2016, 31: 1-10. DOI:10.22203/eCM.v031a01 |
| [20] |
Qi DD, Liu ZH, Wu DS, et al. A study on COMP and CTX-Ⅱas molecular markers for the diagnosis of intervertebral disc degeneration[J]. Biomed Res Int, 2021, 2021: 3371091. |
 2022, Vol.20
2022, Vol.20  Issue(5): 322-326, 333
Issue(5): 322-326, 333


